Testing del Codice con Traffico Reale: Oltre lo Staging con AWS VPC Traffic Mirroring
16 Luglio 2025 - 1 min. read
Alessandro Gallo
DevOps Engineer

Lo sviluppo di un modello di Machine Learning è un processo altamente iterativo, con continui cicli di feedback ottenuti da test e prove precedenti, molto più somigliante ad un esperimento scientifico che ad un progetto di sviluppo.
I Data Scientists sono soliti effettuare molti training su modelli differenti ogni giorno, cercando di trovare quello più robusto per lo scenario su cui stanno lavorando e tenere traccia di tutti i processi svolti si rivela spesso un compito sfidante, anche in un progetto seguito da una singola persona.
Amazon mette a disposizione diversi strumenti per aiutare i Data Scientist a trovare il corretto set di parametri per i loro modelli.
Servizi come Automatic Model Tuning e Amazon SageMaker Autopilot, giungono in aiuto, aiutando ad esplorare velocemente e automaticamente grosse sezioni dello spazio di fase. Tuttavia questi servizi contribuiscono, inevitabilmente, anche alla crescita senza fine di gruppi di parametri per i training e artefatti dei modelli compilati.
Se il progetto è grande abbastanza, sono di solito coinvolti molteplici ingegneri a più livelli. Di conseguenza è fondamentale mantenere un progetto il più strutturato possibile, così come trovare metodologie efficaci per condividere tutti i dataset, i notebook, gli iperparametri e naturalmente i risultati.
I componenti principali di un progetto di Machine Learning, di cui sono necessari versionamenti, indicizzazione e condivisione tra tutti i Data Scientists coinvolti, sono:
Ogni membro del team dovrebbe sempre avere chiaro qualsiasi sia l’ultima versione di ogni componente citato, ed essere così in grado di ricercare velocemente risultati e artefatti di ogni precedente versione dei processi e delle prove effettuate.
Per venire incontro ai Data Scientists nei processi di gestione e strutturazione dei progetti di ML, Amazon ha rilasciato un nuovo servizio: SageMaker Experiments.
Questo nuovo componente di Amazon SageMaker ha come scopo proprio quello di risolvere questa sfida gestionale, fornendo una vista unificata per tutti questi parametri, per i training effettuati e per gli artefatti di output.
In questo articolo, vi presentiamo dunque un caso d’uso reale in cui abbiamo fatto uso estensivo di SageMaker Experiments.
Il progetto aveva come scopo la gestione del clustering di un dataset di clienti molto sparso, contenente diversi milioni di tuple, per estrarre informazioni comportamentali. La struttura del dataset e le feature disponibili avevano reso la scelta dell’algoritmo di clustering e il tuning degli iperparametri tutt’altro che semplice.
Abbiamo testato diversi tipi di algoritmi di clustering (Kmeans, Gaussian mixture, DBSCAN) con differenti combinazioni di feature. Per scoprire inoltre quelle più rilevanti per il clustering stesso abbiamo utilizzato la tecnica PCA e i valori di correlazioni tra le variabili in gioco.
Dopo diverse iterazioni, abbiamo notato che il risultato più stabile si è ottenuto mediante DBSCAN dopo aver effettuato una riduzione di dimensionalità con UMAP (Uniform Manifold Approximation and Projection). L’analisi KNN è servita a trovare il valore ottimo del raggio (eps) per DBSCAN.
UMAP, DBSCAN e KNN sono algoritmi che traggono peraltro grande giovamento dalla parallelizzazione via GPU.
Per garantire la massima efficienza nell’effettuare il clustering del dataset, abbiamo deciso di usare il framework RapidsAI, che include la versione GPU per CUDA di tutti gli algoritmi da noi menzionati e necessari alla nostra pipeline.
AWS offre diversi tagli per le istanze GPU di SageMaker. Per il nostro carico di lavoro abbiamo selezionato una ml.p3.2xlarge per il testing e l’esplorazione dei dati e una ml.g4dn.2xlarge per il training dei modelli.
Per installare RapidsAI, possiamo partire dal sito della libreria. Qui è possibile trovare i comandi di installazione per Conda Python3, che è proprio ciò che ci serviva per la nostra istanza di testing di SageMaker:
conda create -n rapids-0.17 -c rapidsai -c nvidia -c conda-forge \
-c defaults rapids-blazing=0.17 python=3.7 cudatoolkit=10.1
Dopo aver eseguito i comandi, sarà possibile eseguire il nuovo Kernel Jupiter, ma per farlo dovrete chiudere e riaprire il notebook, quindi selezionare “Kernel” -> “Change Kernel”; fatto questo potrete scegliere il nuovo Kernel RapidsAI appena installato.
Nota: ogni volta che si stoppa un’istanza di SageMaker ogni custom Kernel viene perso perchè non fa parte dell’immagine standard che viene associata all’istanza, quindi è necessario procedere di nuovo alla procedura di installazione vista precedentemente.
Alcuni metodi tipici di partizione come K-means o clustering PAM sono particolarmente adatti a trovare cluster sferici o, più in generale, convessi. Di solito funzionano molto bene per cluster compatti e ben separati, pressappoco della stessa dimensione. Di solito sono però sensibili alla presenza di forte anisotropia nei dati (rumore e outlier).
Anche se possono dunque sembrare più che adatti in molte situazioni, ve ne sono molti altri in cui non solo non lo sono, ma in particolare, performano veramente male con:
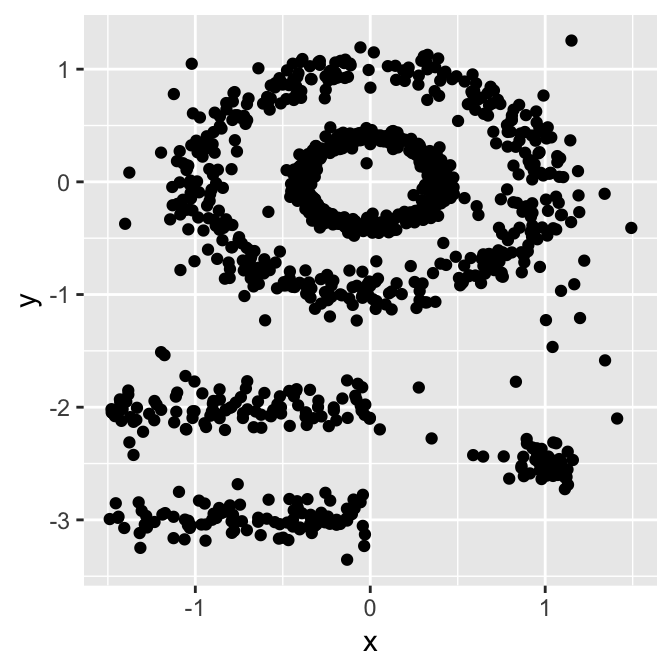
Ed è per questo che abbiamo provato DBSCAN.
Density-based spatial clustering of applications with noise o, in breve, DBSCAN è un algoritmo ben noto di clustering dei dati utilizzato comunemente per i problemi di data mining e machine learning.
Basandosi su un dataset di punti, DBSCAN raggruppa insieme tutti quelli sufficientemente vicini tra di loro rispetto ad una misurazione di distanza, tipicamente Euclidea, con una soglia basata su un numero minimo di punti. È inoltre in grado di marcare come outlier tutti quei punti che risultano particolarmente sparsi, ovvero presenti in regioni a bassa densità.
DBSCAN utilizza principalmente 2 parametri obbligatori:
eps: il raggio locale per gli expanding cluster. Lo si pensi come un valore di STEP - DBSCAN non effettua mai step con valori maggiori di questo, ed indica inoltre quanto vicini debbano essere i punti tra di loro per essere considerati parte di un cluster.
minPoints: il minimo numero di punti vicini tra di loro per formare una regione densa (min. numero di punti nel raggio). Questo di solito è fortemente legato alla dimensione minima del cluster. Per esempio, un valore di 100, significa che abbiamo bisogno di almeno 100 punti per definire un cluster, se i cluster hanno bordi ben definiti e se il dataset non contiene rumore random.
La stima dei parametri è un problema comune a tutti i progetti di ML. Per poter trovare dei valori corretti di eps e minPoints è, di solito, necessario avere una discreta conoscenza della forma del dataset. Di seguito dunque abbiamo alcune considerazioni a carattere generale su come scegliere valori iniziali ragionevoli di eps e minPoints.
eps: se il valore eps scelto è troppo piccolo, gran parte dei dati non verrà raggruppata. Sarà considerato un valore anomalo perché non soddisfa il numero di punti necessario a creare una regione densa. D'altra parte, se il valore scelto è troppo alto, i cluster si uniranno e la maggior parte degli oggetti si troverà nello stesso cluster. L'eps dovrebbe essere scelto in base alla distanza media tra i punti del set di dati (possiamo usare un grafico di k-distance per trovarlo), ma in generale sono preferibili piccoli valori di eps. Va notato che eps è influenzata dalla dimensionality curse, quindi più elementi si hanno, più grande sarà eps, infatti, la distanza euclidea media in un set di dati normalizzato è proporzionale alla dimensionalità.
minPoints: come regola generale, un valore minimo di minPoints può essere derivato da un numero di dimensioni (D) nel set di dati, come minPoints ≥ D + 1. Valori più grandi sono generalmente migliori per set di dati con rumore e formeranno cluster più significativi. Il valore minimo per minPoints deve essere 3, ma maggiore è il dataset, maggiore è il valore minPoints da scegliere.
Al fine di evitare le insidie derivanti dalla dimensionality curse e per ottenere risultati di clustering più stabili ed efficienti, una tecnica comune è quella di utilizzare un algoritmo di riduzione di dimensionalità. Nel nostro caso, abbiamo utilizzato l'analisi di correlazione per la selezione delle feature e quindi la PCA, per trovare quali di queste avessero un impatto più significativo sui componenti principali, risultando quindi buone candidate per essere incluse nel set di dati finale, usato per addestrare il nostro modello.
Tuttavia, l'utilizzo diretto della PCA per la riduzione della dimensionalità ha portato a risultati negativi: il numero di componenti principali necessari a spiegare una percentuale significativa della varianza del dataset è risultato piuttosto alto e, di conseguenza, tutti gli algoritmi di clustering hanno funzionato male.
Le prestazioni PCA non ottimali ci hanno costretto a cercare altrove per la riduzione della dimensionalità. Altri algoritmi comunemente usati a questo scopo sono t-SNE e UMAP.
Entrambi gli algoritmi risultano poco performanti nell'implementazione single-core di Scikit-learn sul nostro dataset molto grande, tuttavia, le cose cambiano radicalmente con l'implementazione GPU CUDA di RapidsAI. Con l'aiuto della GPU, siamo stati in grado di eseguire la riduzione di dimensionalità, per entrambi i metodi, in pochi minuti.
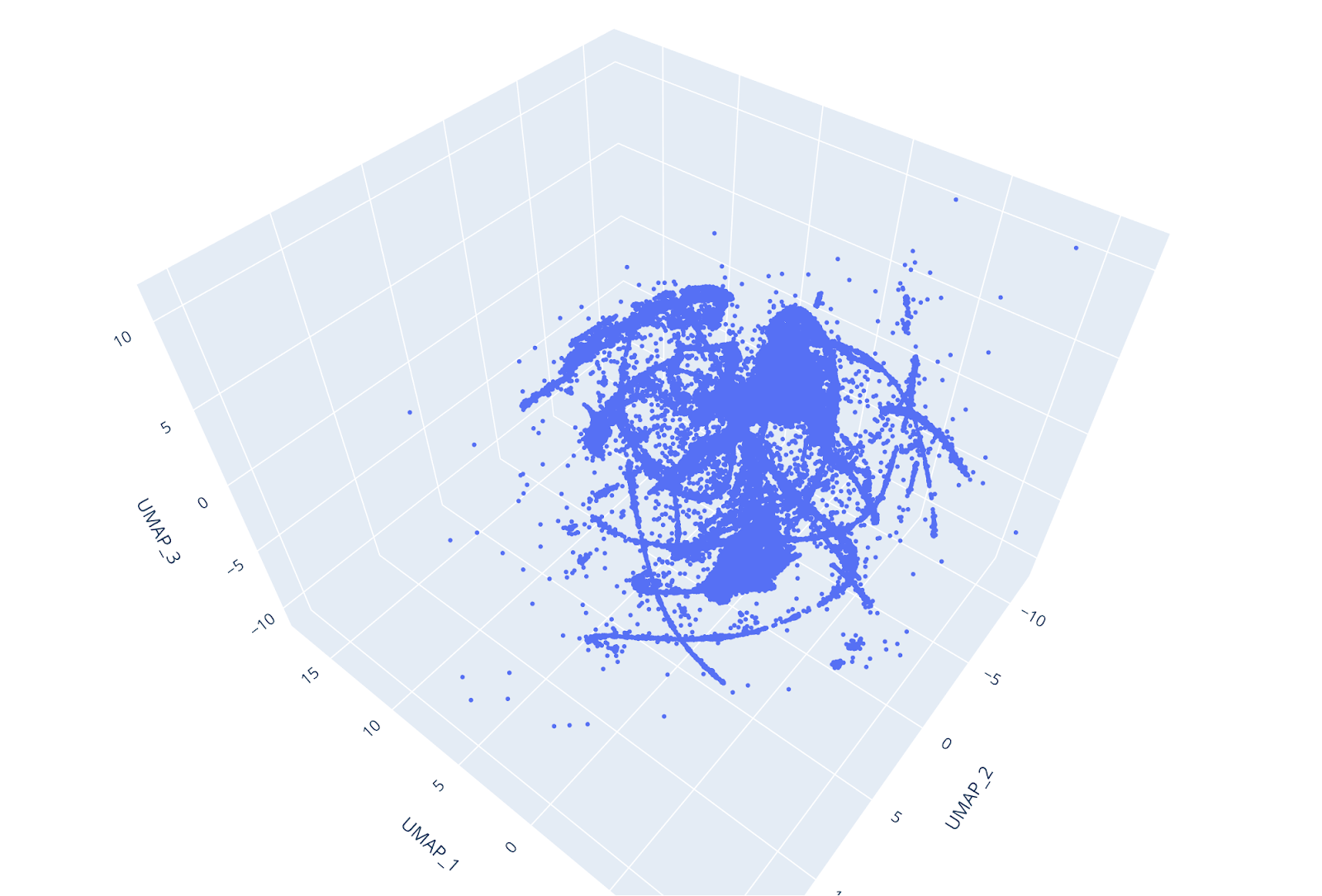
UMAP è più veloce e, nonostante sia un algoritmo di recente scoperta, è generalmente preferibile a t-SNE poiché fa un buon lavoro nel preservare sia la struttura locale che quella globale del set di dati (ecco un esempio).
Inoltre, UMAP non utilizza inizializzazione casuale, quindi i risultati rimangono coerenti tra le diverse esecuzioni. Riducendo il set di dati a 3 dimensioni, i singoli cluster potevano essere facilmente distinti a colpo d'occhio e il punteggio di affidabilità UMAP era molto alto.
UMAP ha una serie di importanti iperparametri che influenzano le sue prestazioni. Questi iperparametri sono:
Misurare le prestazioni di un algoritmo è sempre un passo essenziale in un flusso di lavoro di Machine Learning.
Mentre in un progetto di Machine Learning supervisionato, il modo per farlo è solitamente semplice (addestrare il modello su un sottoinsieme del set di dati e testarlo su un altro sottoinsieme), per un flusso ML non supervisionato, la valutazione oggettiva delle prestazioni è più complicata e ancor di più, se l'esistenza e il numero dei cluster non sono conosciuti né conoscibili a priori.
Un modo comune per risolvere questo problema, consiste nel selezionare una o più metriche obiettivo, legate alle prestazioni dell'output del processo clustering e verificare se, una determinata esecuzione, soddisfa o meno delle soglie target.
Una metrica molto generale, robusta e ampiamente utilizzata è il punteggio Silhouette, che è la metrica che abbiamo selezionato per guidare la nostra valutazione delle prestazioni di clustering.
Il punteggio Silhouette può essere utilizzato per studiare la distanza tra diversi cluster risultanti in un problema, appunto, di clustering; mostra la misura di quanto ogni punto di un cluster sia vicino ai punti dei cluster intorno ad esso e quindi fornisce un modo per valutare visivamente variabili quali il numero di cluster.
Questa misura ha un intervallo compreso tra -1 e 1.
I coefficienti di silhouette vicini a 1 indicano che il campione è lontano dai cluster vicini, quindi buono. Un valore di 0, invece, ci dice che il punto è vicino al confine di decisione tra due cluster vicini. Infine, i valori negativi indicano che i campioni potrebbero essere stati assegnati al cluster sbagliato.
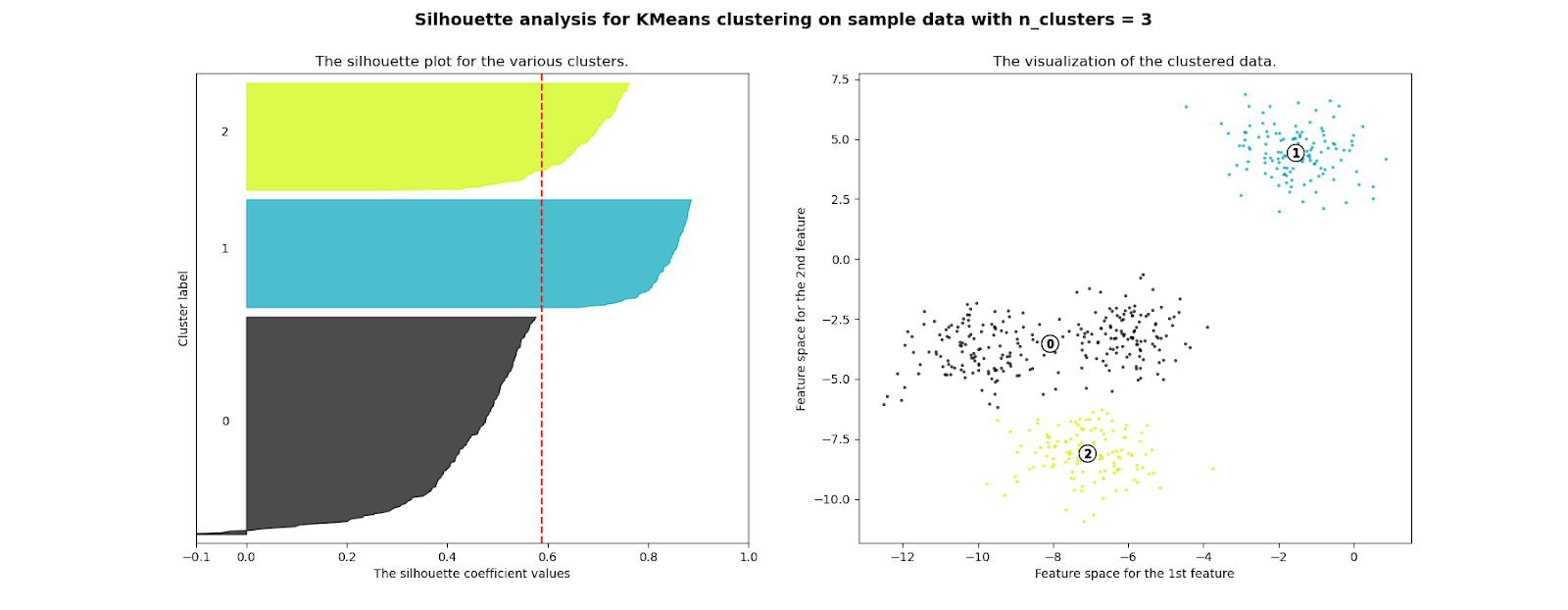
Abbiamo utilizzato questo punteggio per verificare la qualità dei cluster dei nostri clienti e per valutare le prestazioni di diversi algoritmi di clustering e set di iperparametri.
Il completamento del flusso di lavoro di clustering ha richiesto diverse centinaia di iterazioni con diversi algoritmi di pulizia dei dati, euristica, selezione delle funzionalità e clustering.
Amazon SageMaker Experiments aiuta a tenere traccia delle iterazioni sui modelli di ML acquisendo i parametri di input, le configurazioni e i risultati e archiviandoli come "esperimenti". In SageMaker Studio puoi sfogliare esperimenti attivi, cercare quelli precedenti, rivederli insieme ai risultati e confrontare questi risultati.
L'obiettivo di SageMaker Experiments, con la sua API Python, è rendere il più semplice possibile la creazione di tali esperimenti, popolarli con test ed eseguire analisi attraverso prove ed esperimenti.
Puoi eseguire query complesse per trovare rapidamente una versione passata di un test che stai cercando. Puoi anche visualizzare classifiche dei modelli e grafici delle metriche in tempo reale.
Nel corso di questo articolo, verrai guidato attraverso le funzionalità e l'impostazione di SageMaker Experiment attraverso lo scenario del caso d’uso reale che abbiamo affrontato.
Per poter utilizzare SageMaker Experiments, dobbiamo prima accedere a Sagemaker Studio, e per farlo, abbiamo creato alcuni utenti, uno per ogni collaboratore, nella Console AWS.
È necessario disporre delle autorizzazioni corrette per accedere ai servizi di SageMaker tramite la console, se ne si dispone, è sufficiente fare clic sulla barra laterale sinistra della pagina del servizio e accedere al pannello delle proprietà di SageMaker Studio. Lì troverai il pulsante "Add user" per creare un nuovo utente.
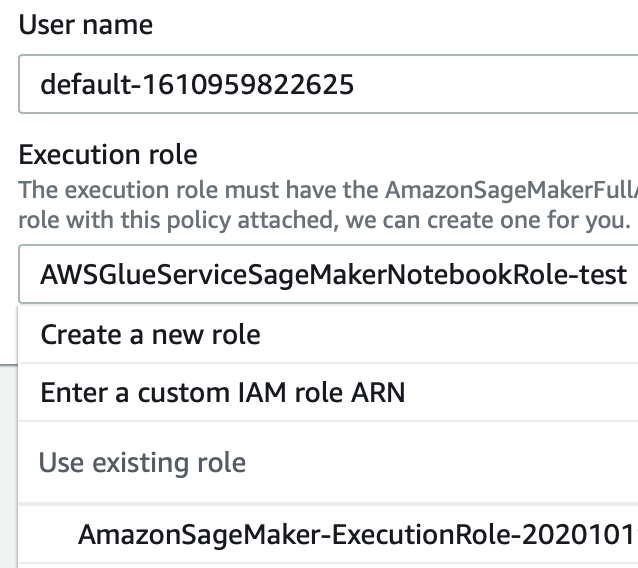
La creazione di un utente è semplice: basta fornire un nome e un ruolo IAM valido, come nell'esempio seguente, e poi si è a posto. Si guardi l'immagine sotto per chiarezza:
Questo è un punto semplice ma cruciale: nella gestione di un progetto, complesso come uno di Machine Learning, sono estremamente importanti i confronti costanti con il cliente. Dandogli la possibilità di valutare facilmente i risultati di ogni esperimento, si rende più facile avere un feedback e si migliorano significativamente le possibilità di un risultato soddisfacente per il progetto.
Prima di iniziare a capire come funziona Experiments, definiamo alcuni concetti chiave che si deve conoscere:
L'obiettivo di SageMaker Experiments è, ovviamente, quello di rendere il più semplice possibile l'accesso e la manipolazione degli esperimenti, popolarli con prove e inoltre fare analisi su test ed esperimenti per prendere decisioni ponderate.
Per aiutarci in questa attività, AWS offre un SDK Python, contenente API di logging e analisi, per integrare Experiments nei notebook.
Prima di iniziare a descrivere il processo in dettaglio, solo una precisazione: abbiamo utilizzato l'approccio più elastico possibile nell'affrontare il nostro problema di Machine Learning con SageMaker, ovvero bring your own container: ciò significa che definiamo il nostro codice ML in script passati ad un Estimator, che viene eseguito su immagini personalizzate inviate ad AWS ECR.
Perché svolgere questo lavoro extra, invece di utilizzare materiale predefinito di AWS? Perché avevamo bisogno di maggiore flessibilità nell'affrontare il nostro problema e, in generale, si vedrà che, a parte casi estremamente rari, probabilmente anche il lettore finirà per fare la stessa cosa con il proprio caso d'uso.
Gli scenari del mondo reale che coinvolgono le aziende hanno condizioni molto specifiche, che possono essere affrontate con maggiore precisione, solo quando gli algoritmi e i modelli sono adattati e messi a punto su quel problema specifico.
In Jupyter Notebooks, gli utenti possono aggiungere automatismi utilizzando le API di SageMaker; quando si definisce un Estimator per il training di un modello, è possibile aggiungere una definizione di Esperiments, in questo modo fondamentalmente colleghiamo l'operazione a una tupla nella console di Experiments in SageMaker Studio.
Per il nostro processo di clustering questa possibilità era importante, perché avevamo l'esplicita necessità di fornire aggiornamenti settimanali sulle prestazioni dei nostri modelli. Di solito questa è, come detto, una buona pratica per aiutare a creare una chiara comprensione del problema con il cliente e, in generale, per evitare confusione.
Prima di passare un esperimento a uno stimatore, come spiegato nella documentazione ufficiale di AWS, dobbiamo generarlo da zero; in una nuova cella di un Jupyter Notebook scriviamo queste righe:
import sagemaker
from smexperiments.experiment import Experiment
from sagemaker import get_execution_role
from sagemaker.session import Session
experiment_name="my-TestExperiment"
role = get_execution_role()
session=Session()
sm_client=session.boto_session.client('sagemaker')
experiment = None
for exp in Experiment.list():
if exp.experiment_name==experiment_name:
experiment = Experiment.load(experiment_name=experiment_name,sagemaker_boto_client=sm_client)
print(f"Experiment {experiment_name} is loaded!")
break;
if experiment == None:
experiment = Experiment.create(
experiment_name = experiment_name,
description = "My Test Experiment",
tags = [{'Key': 'project', 'Value': 'customer-segmentation'}]
)
print(f"Experiment {experiment_name} is created!")
In queste righe di codice stiamo descrivendo un nuovo esperimento e non solo: se l'esperimento esiste già nella lista degli esperimenti, che dipende dalle credenziali dell'utente loggato su Jupyter, quell'esperimento viene caricato, invece di crearne uno nuovo.
Perché l'abbiamo fatto? Perché volevamo dividere gli esperimenti per categorie, quindi utilizzare le prove, per raggruppare diversi Training Job. In questo modo si possono mantenere le cose più pulite.
È quindi possibile aggiungere un esperimento a uno stimatore, ma prima di farlo, è necessario creare dei Trial e aggiungerli all'esperimento stesso (ecco perché possiamo caricare anche un esperimento esistente).
I Trial sono gruppi in cui ogni evento di training viene mantenuto. Sono definiti in un esperimento e possono essere utilizzati per dividere e mantenere diversi Training Job, con diversi set di parametri, separati e puliti.
Noi lo abbiamo fatto perché abbiamo provato un gran numero di set di iperparametri per mettere a punto i modelli, in parte anche perché avevamo un set di dati iniziale molto scarso, che era molto difficile da analizzare in termini di clustering.
Per aggiungere un Trial a un esperimento SageMaker, ecco un codice di esempio da utilizzare in una cella sotto quella con l'inizializzazione dello stesso.
from smexperiments.trial import Trial
from smexperiments.trial_component import TrialComponent
from smexperiments.tracker import Tracker
import time
from time import strftime
trial_create_date = strftime("%Y-%m-%d-%H-%M-%S")
trial_name ="test-trial-{}".format(trial_create_date)
trial = None
try:
trial = Trial.load(trial_name, sagemaker_boto_client=sm_client)
print(f"Trial {trial_name} is loaded!")
except:
trial = Trial.create(trial_name = trial_name,
experiment_name = experiment.experiment_name,
tags = [{'Key': 'my-experiments', 'Value': 'trial 1'}])
print(f"Trial {trial_name} is created!")
In questo caso, per poter distinguere tra loro i vari Trial, ne generiamo dinamicamente il nome: una pratica comune; si può fare a seconda della categoria di un Trial o dei parametri coinvolti. Basta essere consapevoli del limite di caratteri del Trial (120 caratteri), quindi non rendiamo il nome troppo lungo ed evitiamo i caratteri speciali in quanto non sono consentiti.
Ora che abbiamo aggiunto un Trial ad un esperimento, è possibile aggiungere quest’ultimo ad uno stimatore per renderlo disponibile dall'IDE di SageMaker Studio.
Gestiscono i Training Job e le distribuzioni end-to-end dei task di deploy di Amazon SageMaker. È una classe Python che definisce tutte le caratteristiche di un training. Per aggiungere il supporto ad Experiments è sufficiente aggiungere una riga di codice in più alla definizione di uno stimatore come nell'esempio seguente:
estimator.fit(
inputs={"train":sagemaker.session.s3_input(s3_data=data_location)},
wait=True,
logs='All',
experiment_config={
"ExperimentName":experiment.experiment_name,
"TrialName" : trial.trial_name,
"TrialComponentDisplayName" : "Training",
}
)
Fondamentalmente, quando eseguiamo il metodo .fit() per avviare un Training Job, SageMaker riconosce il parametro experiment_config e collega quel particolare Job alla dashboard di Sagemaker Studio Experiment.
Ciò dimostra quanto sia semplice creare un esperimento, ma è anche necessario definire un Tracker, al fine di inviare effettivamente le informazioni alla dashboard dell'esperimento.
Per inviare in modo efficace le informazioni alla dashboard di SageMaker Studio, è necessario configurare un tracker. Un tracker è fondamentalmente un logger che accetta diversi input e li invia a schede specifiche della console di Experiments, a seconda del metodo utilizzato.
Grazie al tracker e ad alcuni log predefiniti offerti da SageMaker Experiments, si può ottenere una descrizione particolarmente completa del proprio test come nell'immagine sottostante:
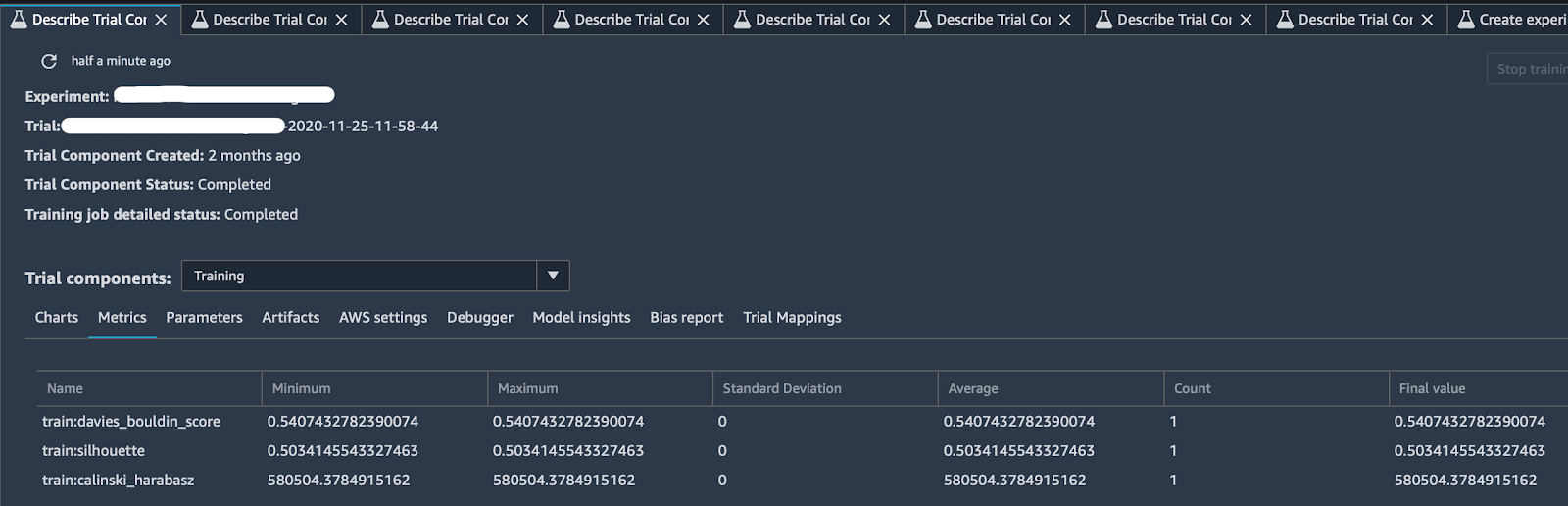
Facendo clic sulla scheda "Describe Trial Component", si può cliccare su qualsiasi intestazione di colonna per visualizzare le informazioni su ciascun componente di un Trial, in particolare:
Un Tracker può, e generalmente deve, essere aggiunto ad uno script per l’Estimator, che contiene la propria logica di Machine Learning.
Dopo aver definito i parametri di ingresso (gli argomenti dello script), si può aggiungere un Tracker in questo modo:
# Loading Sagemaker Experiments Training Tracker tracker=tracker.Tracker.load()
Dopo questa linea di codice è possibile cominciare ad inviare informazioni a SageMaker Studio. Nel nostro caso abbiamo utilizzato 3 metodi del tracker:
tracker.log_parameters({ "param1": NUM, "param2": NUM, ...})Che è utilizzato per inviare parametri extra alla scheda “Parameters” nella dashboard di quel particolare trial.
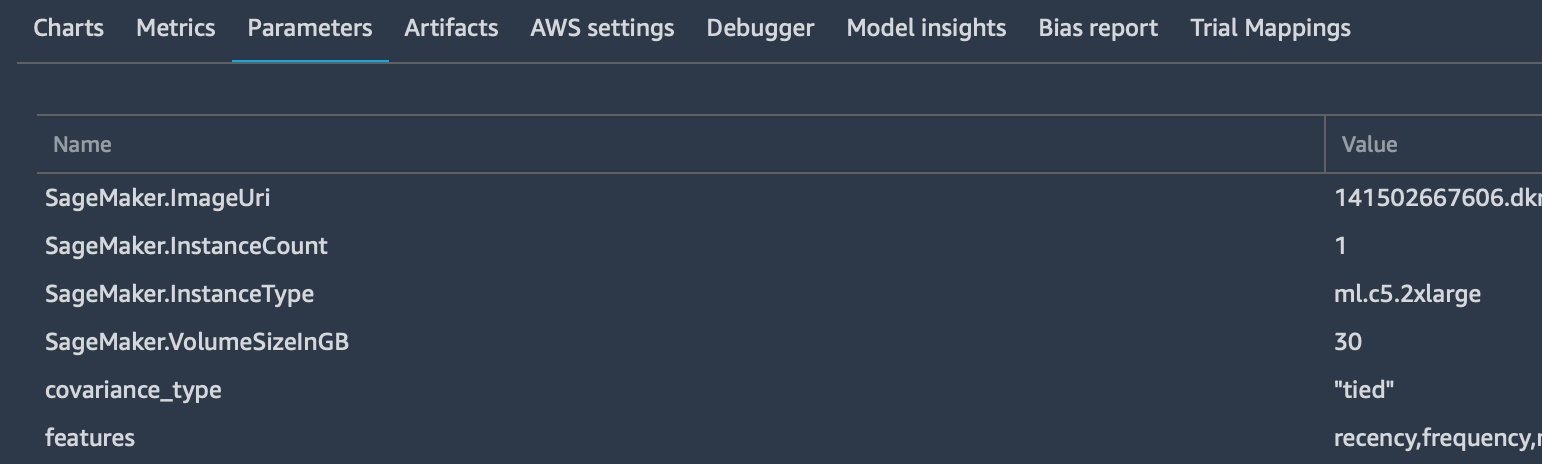
tracker.log_input(“key”, “value”, media_type=TYPE)
Viene utilizzato per registrare tutti i parametri di input desiderati, specificando anche il media type per consentirne l'anteprima, se possibile, nel browser. Questi parametri verranno aggiunti alla scheda "Metrics":
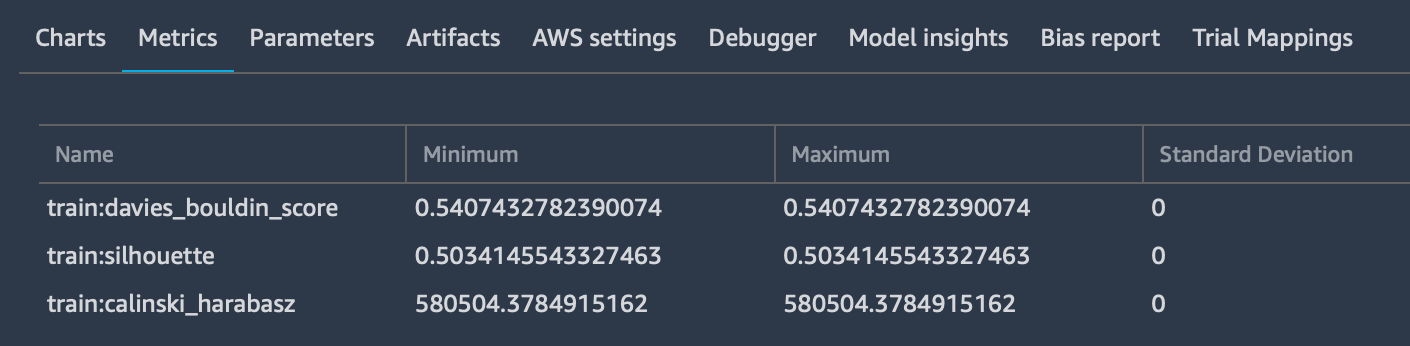
tracker.log_output(name="Some link name", media_type="s3/uri", value=s3_uri)
Si usa per inviare output specifici alla scheda “Artifacts” che contiene già le path dei bucket di storage di Amazon S3 sia per l’input dataset che per l’output model.
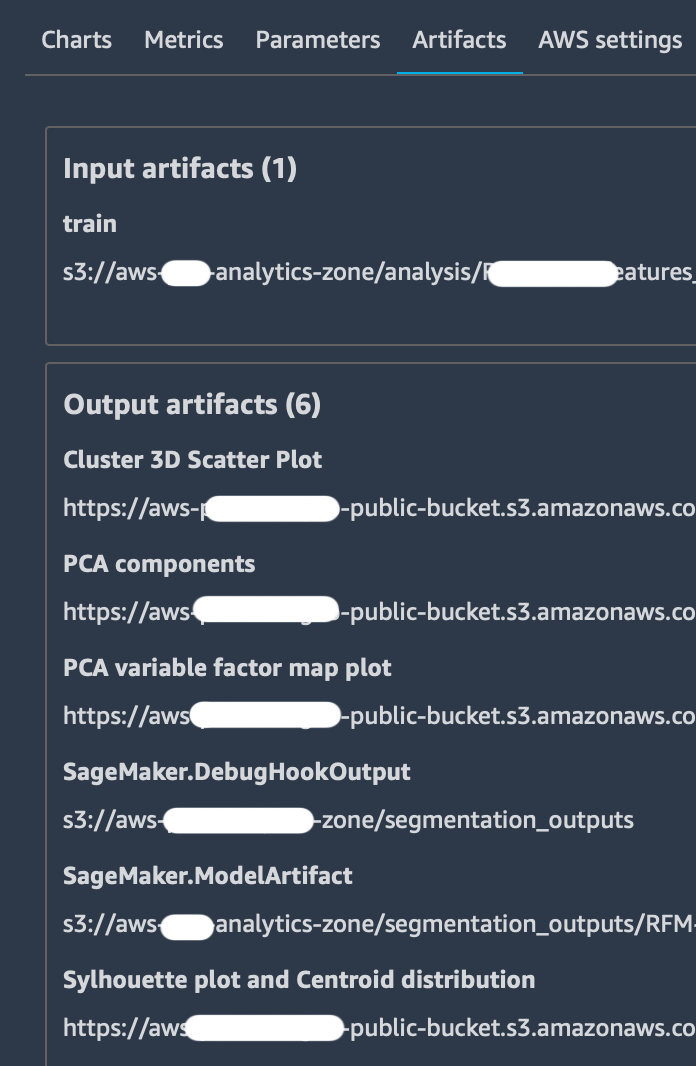
Infine ricordarsi di chiudere sempre il tracker prima di concludere lo script.
tracker.close()
Come indicato anche dalla documentazione di AWS, si possono confrontare esperimenti, Trial e componenti dei Trial, aprendoli nella Studio Leaderboard. In questa schermata puoi eseguire le seguenti azioni:
Quando hai uno o più esperimenti, puoi aprire la Leaderboard per confrontare i risultati. Scegli gli esperimenti o le prove che desideri confrontare; fai clic con il tasto destro su uno degli elementi, quindi su "Open in trial component list". Si aprirà la Leaderboard e ti verrà presentato un elenco di Esperiment associati, come mostrato nell'immagine seguente:
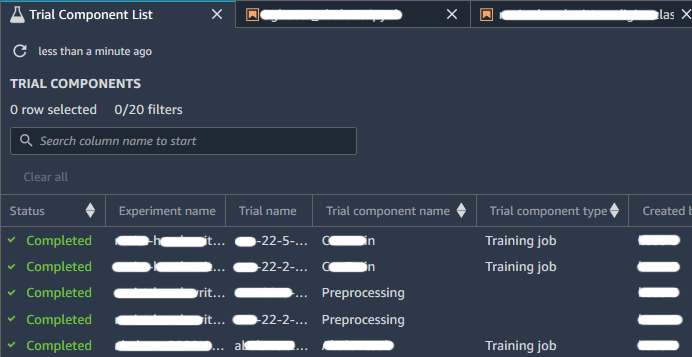
Dalla nostra sperimentazione abbiamo scoperto che il Tracker può essere utilizzato solo quando l'Estimator viene avviato in modalità online, ovvero specificando un tipo di istanza che non è "locale".
Significa eseguire il training su un'istanza appena creata, invece di eseguirlo direttamente sull'istanza in cui risiede il notebook.
In questo articolo, abbiamo visto come sfruttare le nuove funzionalità offerte da Amazon SageMaker Experiments per gestire il clustering di una notevole quantità di dati.
Nel nostro viaggio insieme a voi, abbiamo utilizzato queste nuove funzionalità per condividere meglio i risultati nel nostro team e con il cliente e per coordinare agevolmente gli sforzi degli ingegneri coinvolti.
Non siamo andati troppo in profondità con le caratteristiche di SageMaker Experiment, ma abbiamo condiviso con voi i tratti più importanti in quanto vorremmo incoraggiare il lettori a provare e sperimentare da soli.
Lungo il percorso descritto, abbiamo anche visto come utilizzare due interessanti algoritmi di clustering: DBSCAN e UMAP. Questi sono particolarmente adatti per trovare gruppi di dati simili quando il set di dati originale è scarso e disomogeneo, ma richiedono un'istanza abilitata CUDA.
Infine, abbiamo anche discusso brevemente il concetto chiave di "bring your own container" in SageMaker che abbiamo trovato fondamentale per completare la nostra analisi: come abbiamo detto, quando si ha a che fare con scenari di vita reale complessi, algoritmi preconfezionati offerti da AWS SageMaker non sono, di solito, abbastanza flessibili.
Per concludere speriamo che la lettura vi sia piaciuta, per qualsiasi domanda non esitate a contattarci, per parlare di Machine Learning o di qualsiasi altro argomento relativo al fantastico mondo del Cloud Computing!
Restate sintonizzati per altri articoli sul Machine Learning. A tra 14 giorni qui su #Proud2beCloud!